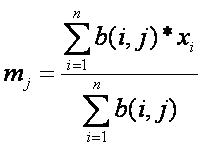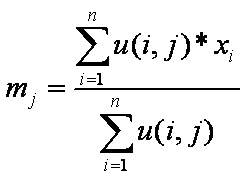El algoritmo de k-medias.
Suponga que tenemos n características
en vectores x1, x2, ..., xn, donde cada x, está representado en un espacio m
dimensional y sabemos que están agrupados en k cúmulos (k<n). Definimos
mj como la media
del j-ésimo cúmulo. Si los cúmulos están bien separados, podemos usar una
mínima distancia de clasificación para separarlos. Esto es, podemos decir que xi esta en el j-ésimo cúmulo si || xi
– mj || es el mínimo con respecto a los k cúmulos. Esto sugiere
el siguiente algoritmo para encontrar la k medias:
·
Haga una estimación inicial para la k
medias m1, m2, ..., mk.
·
Mientras no cambie alguna media:
o
Use la media estimada para clasificar
los datos en cúmulos. b(i,j) = 1 si el
i-ésimo dato, es el más cercano a la j-ésima media.
o
Para cada uno de los cúmulos
§
Calcule la nueva media mi, utilizando la nueva clasificación
o
fin.
·
fin.
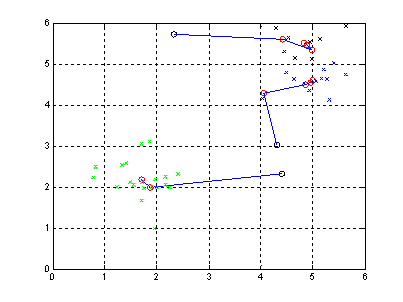
Aquí tenemos un ejemplo que muestra las dos medias
como se mueven en el procedimiento para calcular las medias.
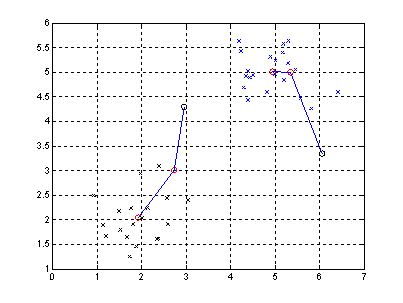
Este es un ejemplo simple del algoritmo de k-medias.
Este puede ser visto como un algoritmo voraz para particionar en k cúmulos, el
cual la suma de las distancias al cuadrado, al centro de caca cúmulo. Pero este
algoritmo tiene algunas debilidades.
·
La manera de inicializar no se especifica. Una forma
común es comenzar seleccionado aletoriamento k medias de los ejemplos.
·
Los resultados dependerán del valor inicial de las medias
y frecuentemente pasa que particiones subóptimas son encontradas. La solución
estándar es calculada atizando diferentes puntos de arranque.
·
Puede pasar que un conjunto de ejemplos cercano a una
media este vacío, por lo que no puede ser modificada. Esto es un inconveniente
del método y debe ser manejado en la implementación, pero puede ser ignorado.
·
Los resultados depende de la métrica utilizada para medir
|| x - mi
||. Una solución popular es normalizar cada variable por la desviación
estándar, aunque no siempre es deseable.
·
La solución de pende del número de cúmulos seleccionado.
El ultimo problema es particularmente
pesado, normalmente no tenemos manera de saber cuantos cúmulos existen.
En el ejemplo mostrado enseguida, el conjunto de datos es inicializado
utilizando 3 medias. ¿Esto es mejor o peor que cuando se utilizando únicamente
2 medias? La respuesta puede solo encontrarse utilizando diferentes
clasificadores o uniendo algunas subclases en un procedimiento posterior.
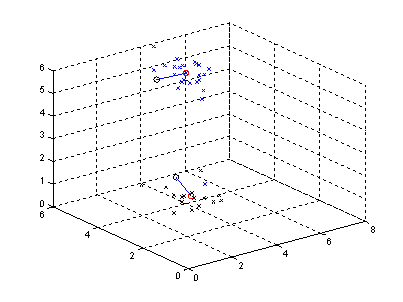
El algoritmo puede calcular medias en cualquier dimensión, a
continuación se presenta un ejemplo del algoritmo de k medias aplicado en tres
dimensiones.
A continuación se da el código para hacer el llamado a una función
implementada en MATLAB, para llamar a la función de kmedias.
clear;
for n = 1 :40
if(unifrnd(0,1) > 0.5)
x(n) = normrnd(2, 0.5);
y(n) = normrnd(2, 0.5);
z(n) = normrnd(2, 0.5);
else
x(n) = normrnd(5, 0.5);
y(n) = normrnd(5, 0.5);
z(n) = normrnd(5, 0.5);
end;
end
datos = [x', y', z'];
%datos = [x', y'];
[medias, b ] = kmeans(datos, 2)
dim = length(datos(1, :));
for k=1:length(x)
switch(b(k))
case 1
if (dim == 2)
plot(datos(k,1), datos(k,2), 'kx'), end;
if (dim == 3)
plot3(datos(k,1), datos(k,2), datos(k,3), 'kx'), end;
case 2
if (dim == 2)
plot(datos(k,1), datos(k,2), 'bx'), end;
if (dim == 3)
plot3(datos(k,1), datos(k,2), datos(k,3), 'bx'), end;
case 3
if (dim == 2)
plot(datos(k,1), datos(k,2), 'gx'), end;
if (dim == 3)
plot3(datos(k,1), datos(k,2), datos(k,3), 'gx'), end;
end;
end;
hold off;
La función para calcular la k-medias es:
function [medias, b] = kmeans(datos, num_medias)
ndatos = length(datos(:,1));
dim = length(datos(1,:));
if (dim == 2) plot(datos(:,1), datos(:,2), 'x'), end;
if (dim == 3) plot3(datos(:,1), datos(:,2), datos(:,3), 'x'), end;
hold on;
grid on;
medias = zeros(num_medias, dim);
np = zeros(num_medias, dim);
for n = 1: num_medias
for m = 1:dim
medias(n,m) = unifrnd(
min(datos(:, m)), max(datos(:, m)) );
end;
end;
medias_old = medias;
if (dim == 2) plot(medias(:,1), medias(:,2), 'ko'); end;
if (dim == 3) plot3(medias(:,1), medias(:,2), medias(:,3), 'ko'), end;
for iter = 1 :100
for k = 1: ndatos
dist =
distancia(datos(k,:), medias(1, :));
b(k) = 1;
for ii = 2 :num_medias
aux = distancia(datos(k,:),
medias(ii, :));
if( dist > aux)
b(k) = ii;
dist = aux;
end;
end;
end;
medias = zeros(num_medias, dim);
np = zeros(num_medias,
dim);
for k = 1 : ndatos
n = b(k);
for m = 1: dim
medias(n, m ) = medias(n,m) +
datos(k, m);
np(n,m) = np(n,m) +1;
end;
end;
for n=1: num_medias
for m = 1 :dim
if( np(n,m) ~= 0)
medias(n, m) =
medias(n,m) /np(n,m);
else
medias(n,m) = 0;
end;
end;
end;
if (dim == 2)
plot(medias(:,1), medias(:,2), 'ro'), end;
if (dim == 3) plot3(medias(:,1),
medias(:,2), medias(:,3),'ro'), end;
if (max(max(abs(medias_old -
medias)))<0.01)
break;
end;
for n = 1: num_medias
if (dim == 2)
plot([medias(n,1), medias_old(n,1)],[medias(n,2), medias_old(n, 2)]), end;
if (dim == 3) plot3([medias(n,1),
medias_old(n,1)],[medias(n,2), medias_old(n, 2)],[medias(n,3), medias_old(n,
3)] ), end;
end;
medias_old = medias;
end;
function d = distancia(dx,
media)
d = 0;
n = length(dx);
for k = 1: n
d = d + (dx(k) - media(k))^2;
end;
d = d^0.5;
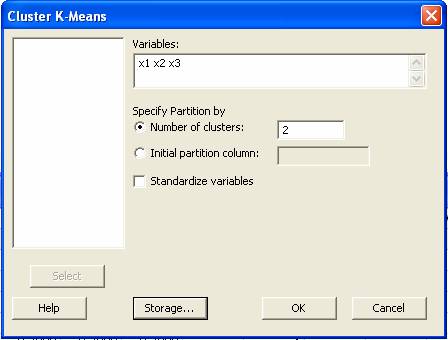
Algoritmo de K-medias en MINITAB
Entrar al menú
Stat > Multivariate >
Cluster K-Means
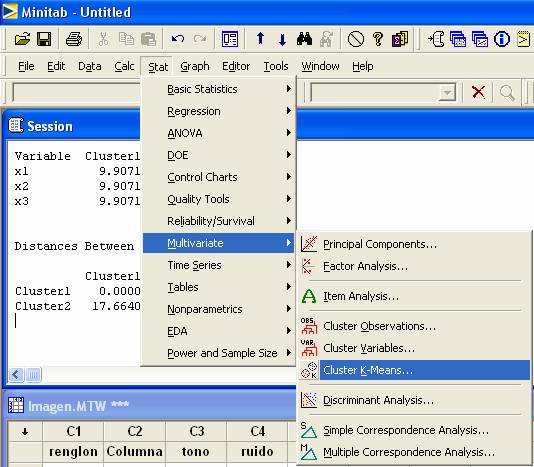
Después
aparecerá la caja de dialogó
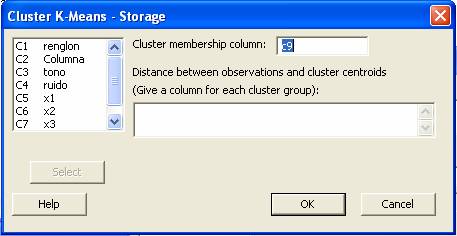
En
la opción Storage, espeficar la columna donde quedará la etiqueta de
pertenencia, en una caja de dialogo como la siguiente.
Ejemplo 1.
Tenemos
la información nutricional de 12 cereales, tales como, proteínas,
carbohidratos, grasas, calorías y Vitamina A. Deseamos hacer un análisis de
cúmulos para saber que cereales tienen propiedades similares.
|
Cereal |
Proteínas |
Carbohidratos |
Grasas |
Calorías |
vitamina A |
|
Life |
6 |
19 |
1 |
110 |
0 |
|
Grape Nuts |
3 |
23 |
0 |
100 |
25 |
|
Super Sugar Crisp |
2 |
26 |
0 |
110 |
25 |
|
Special K |
6 |
21 |
0 |
110 |
25 |
|
Rice Krispies |
2 |
25 |
0 |
110 |
25 |
|
Raisin Bran |
3 |
28 |
1 |
120 |
25 |
|
Product 19 |
2 |
24 |
0 |
110 |
100 |
|
Wheaties |
3 |
23 |
1 |
110 |
25 |
|
Total |
3 |
23 |
1 |
110 |
100 |
|
Puffed Rice |
1 |
13 |
0 |
50 |
0 |
|
Sugar Corn Pops |
1 |
26 |
0 |
110 |
25 |
|
Sugar Smacks |
2 |
25 |
0 |
110 |
25 |
La
solución en MINITAB es
|
Cereal |
Proteina |
Carbo |
Grasa |
Calorias |
VitaminA |
clase |
|
Life |
6 |
19 |
1 |
110 |
0 |
1 |
|
Grape Nuts |
3 |
23 |
0 |
100 |
25 |
3 |
|
Super Sugar Crisp |
2 |
26 |
0 |
110 |
25 |
3 |
|
Special K |
6 |
21 |
0 |
110 |
25 |
4 |
|
Rice Krispies |
2 |
25 |
0 |
110 |
25 |
3 |
|
Raisin Bran |
3 |
28 |
1 |
120 |
25 |
6 |
|
Product 19 |
2 |
24 |
0 |
110 |
100 |
5 |
|
Wheaties |
3 |
23 |
1 |
110 |
25 |
3 |
|
Total |
3 |
23 |
1 |
110 |
100 |
5 |
|
Puffed Rice |
1 |
13 |
0 |
50 |
0 |
2 |
|
Sugar Corn Pops |
1 |
26 |
0 |
110 |
25 |
3 |
|
Sugar Smacks |
2 |
25 |
0 |
110 |
25 |
3 |
El algoritmo de k medias difusas.
Este algoritmo para cálculo de medias es algunas
veces llamado duro o crespo, porque cada característica del vector x es al mismo tiempo miembro y no
miembro de un cúmulo en particular. Esto es un cúmulo difuso o suave, en el
cual cada característica del vector x puede tener un grado de membresía en cada
cúmulo. El algoritmo de k medias difusas de Dunn y Bezdek es :
·
Haga una estimación inicial para la k
medias m1, m2, ..., mk.
·
Mientras no cambie alguna media:
o
Use la media estimada para encontrar el
grado de membresía u(i,j) de el i-ésimo
dato xi, en el j-ésimo cúmulo; por ejemplo, si a(i,j) = exp(- || xi – mj ||2 ), uno puede
usar u(i,j) = a(i,j) / sum_i a(i,j)
o
Para cada uno de los cúmulos
§
Calcule la nueva media mj, utilizando el
grado de membresía;
o
fin.
·
fin.
Ejemplo 2
Repetir el ejemplo de la imagen y
calcular el cúmulo al que pertenece cada píxel para diferentes niveles de
ruido.
Función Discriminante.
Asumiendo que nuestros
datos x, en un espacio de dimensión D, siguen una distribución normal
multidimencional dada por
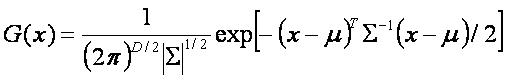
donde m es la media y S la
matriz de covarianza.
Definimos
La verosimilitud de un dato dado una distribución
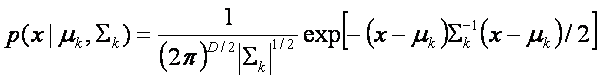
La probabilidad a
posteriori utilizando la regla de Bayes, la cual nos da la probabilidad de que
la k-ésima distribución genero el
dato x, como
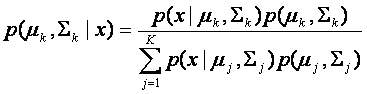
![]()
y la probabilidad a priori p(mk,Sk)
Si asumimos que la probabilidad
a priori esta dada por una constante (es la misma para todos los valores)
entonces podemos reescribir la probabilidad a posteriori como
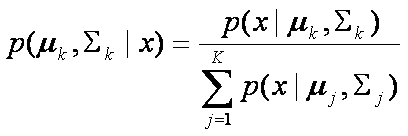
que resulta ser la
verosimilitud normalizada.
Un criterio de clasificación
estipula que la observación x
pertenece a la k-ésima clase si y solo si
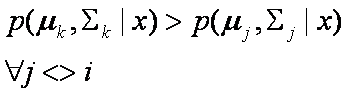
Si consideramos el caso en
el cual tenemos la misma varianza y sacando logaritmo natural a ambos lados de
la ecuación tenemos
(x-mk)TSk-1(x-mk)+ ln(cte) > (x-mj)TSj-1(x-mj) ln(cte)
(x-mk)T (x-mk) > (x-mj)T (x-mj)
xTx-2mkTx+mkT mk > xTx-2mjTx+mjT mj
-2mkTx-mkT mk > -2mjTx+mjT mj
mkx+bk>
mjx+bj
Lo cual nos lleva a una
función discriminante lineal dada por mkx+bk=y.
Si evaluamos esta función podemos decir para un nuevo valor x cual es la clase a la que pertenece.
Para llevar a cabo esta
opción en MINITAB hacemos:
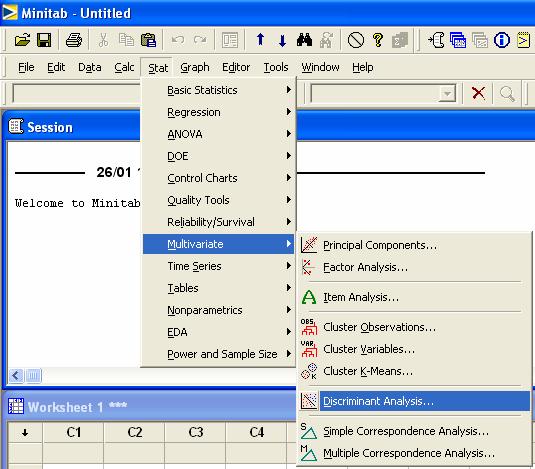
Después aparecerá la caja
de diálogo
En la caja de grupos se coloca
la columna en donde se encuentra la clasificación dada. En la caja de
Predoctores se dan las columnas correspondientes a los valores que caracterizan
cada objeto clasificado.
Ejemplo 3
Considere el caso donde
existen dos clases. La clase w1 = {2, 2.01, 1.99} y la clase w2 = {3.0, 2.99,
3.01}. Determinar la función discriminante para cada una de las clases
Discriminant Analysis: clase versus
valor
Linear Method for Response: clase
Predictors: valor
Group 1 2
Count 3 3
Summary of classification
True Group
Put into Group 1 2
1 3 0
2 0 3
Total N 3 3
N correct 3 3
Proportion 1.000 1.000
N = 6 N Correct = 6 Proportion Correct = 1.000
Squared Distance Between Groups
1 2
1 0.0 10000.0
2 10000.0 0.0
Linear Discriminant Function for Groups
1 2
Constant -20000
-45000
valor 20000 30000
La desigualdad de funciones
discriminante resultante es
-2000 + 2000 valor
> -4500 + 3000 valor
|
valor |
F1 |
f2 |
f1>f2 |
|
… |
… |
… |
… |
|
2 |
2000 |
1500 |
1 |
|
2.1 |
2200 |
1800 |
1 |
|
2.2 |
2400 |
2100 |
1 |
|
2.3 |
2600 |
2400 |
1 |
|
2.4 |
2800 |
2700 |
1 |
|
2.5 |
3000 |
3000 |
* |
|
2.6 |
3200 |
3300 |
0 |
|
2.7 |
3400 |
3600 |
0 |
|
2.8 |
3600 |
3900 |
0 |
|
2.9 |
3800 |
4200 |
0 |
|
3 |
4000 |
4500 |
0 |
|
… |
… |
… |
… |
Si rescribimos la función
tenemos
4500-2000 + (2000-3000) valor > 0
2500 – 1000 valor > 0
|
valor |
f1-f2 |
|
… |
… |
|
2 |
500 |
|
2.1 |
400 |
|
2.2 |
300 |
|
2.3 |
200 |
|
2.4 |
100 |
|
2.5 |
0 |
|
2.6 |
-100 |
|
2.7 |
-200 |
|
2.8 |
-300 |
|
2.9 |
-400 |
|
3 |
-500 |
|
… |
… |
Si igualamos a cero la
función, podemos encontrar de manera analítica la frontera entre clases así
2500 – 1000 valor = 0
valor = 2500/1000
valor = 2.5
En el caso de una sola
dimensión tenemos que la frontera resulta ser una constante, en dos dimensiones
es una línea recta, en tres dimensiones un plano y en general para más de tres
dimensiones es un hiperplano.
Ejemplo 4
Con
el propósito de regular la pesca de salmón es deseable identificar cuando su
origen en Alaska o Canada. Cincuenta
peses de origen fueron pescados y los anillos de crecimiento fueron
medidos cuando ellos vivían en agua fresca y cuando vivían en agua salada. La
meta es estar en posibilidades de identificar si nuevos individuos pescados
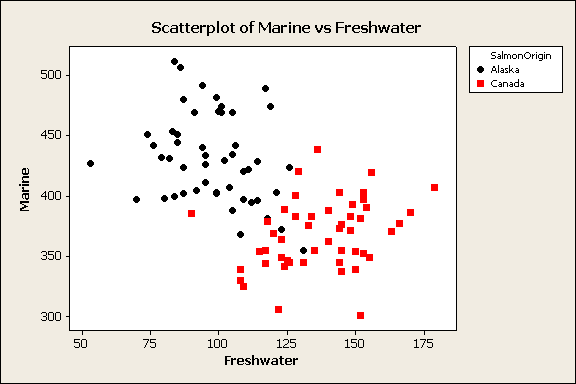
provienen de Alaska o Canada. Los datos para procesar se encuentran en el
archivo salmon.mtw y la siguiente figura muestra una grafica de cómo se
distribuyen.
a) Calcular las funciones discriminantes lineales de cada
clase
b) La función lineal que marca la frontera entre clases
Solución
Discriminant
Analysis: SalmonOrigin versus Freshwater, Marine
Linear Method for Response: SalmonOrigin
Predictors: Freshwater, Marine
Group Alaska Canada
Count 50 50
Summary of classification
True Group
Put into Group Alaska Canada
Alaska 44 1
Canada 6 49
Total N 50 50
N correct 44 49
Proportion 0.880 0.980
N = 100 N Correct =
93 Proportion Correct = 0.930
Squared Distance Between Groups
Alaska Canada
Alaska 0.00000 8.29187
Canada 8.29187 0.00000
Linear Discriminant Function for Groups
Alaska Canada
Constant -100.68 -95.14
Freshwater 0.37 0.50
Marine 0.38 0.33
Summary of Misclassified Observations
Squared
Observation True Group Pred Group
Group Distance Probability
1** Alaska Canada
Alaska 3.544 0.428
Canada 2.960 0.572
2** Alaska Canada
Alaska 8.1131 0.019
Canada 0.2729 0.981
12** Alaska Canada
Alaska 4.7470 0.118
Canada 0.7270 0.882
13** Alaska Canada
Alaska 4.7470 0.118
Canada 0.7270 0.882
30** Alaska
Canada Alaska
3.230 0.289
Canada 1.429 0.711
32** Alaska Canada
Alaska 2.271 0.464
Canada 1.985 0.536
71** Canada Alaska
Alaska 2.045 0.948
Canada 7.849 0.052
a) La función lineal discriminante es
-100.68 + 0.37 Freshwater + 0.38 Marine > -95.14 + 0.5 Freshwater +
0.33 Marine
-5.54 -0.13 Freshwater + 0.05 Marine > 0
b) Si igualamos a cero y despejamos el valor de Marine
de la ecuación anterior tenemos
-5.54 -0.13 Freshwater
+ 0.05 Marine = 0
Marine = (5.54 + 0.13
Freshwater)/ 0.05
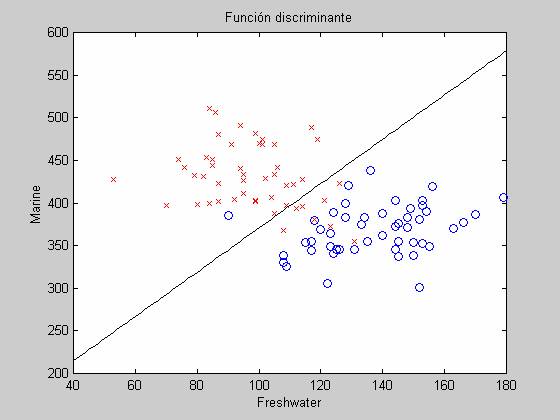
En la siguiente figura en rosa,
aparece los puntos de la línea correspondiente a la ecuación Marine = (5.54 +
0.13 Freshwater)/ 0.05, que es donde la función discriminante es igual a cero.
Los puntos en azul corresponden a los datos dados.
Ejemplo 5
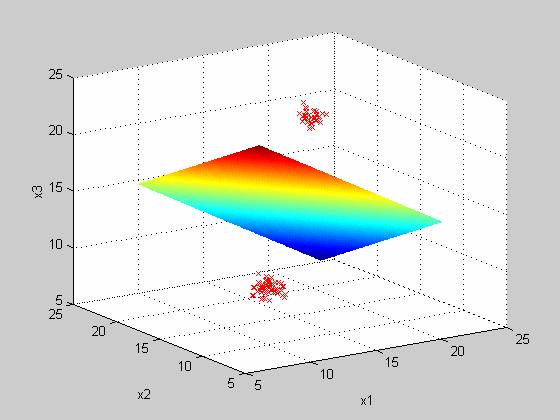
Una
de las aplicaciones del análisis de cúmulos es el filtrado y segmentación de
imágenes. Las imágenes son representadas por arreglos bidimensionales, cada
punto (píxel) almacena la información del tono de gris tal como se muestra en
la siguiente imagen.
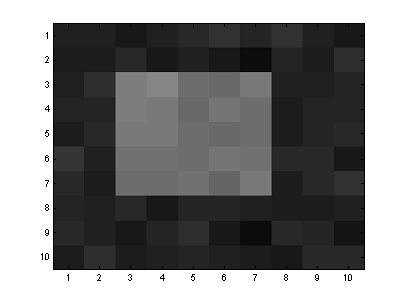
a) Graficar los datos correspondientes
a la figura con un nivel de ruido sigma = 0.5
b) Para la información contenida en el
archivo imagen.mtw, calcular la función discriminante
Discriminant
Analysis: tono versus x1, x2, x3
Linear Method for Response: tono
Predictors: x1, x2, x3
Group 10 20
Count 64 36
Summary of classification
True Group
Put into Group 10 20
10 64 0
20 0 36
Total N 64
36
N correct 64 36
Proportion 1.000 1.000
N = 100 N Correct =
100 Proportion Correct = 1.000
Squared Distance Between Groups
10 20
10 0.00 1142.29
20 1142.29 0.00
Linear Discriminant Function for Groups
10 20
Constant -570.9
-2284.1
x1 35.1 70.0
x2 27.7 55.7
x3 51.3 102.6
c) Graficar la frontera entre las
clases dadas
-570.9 + 35.1 x1 + 27.7 x2 + 51.3 x3 >
-2284.1 + 70 x1 + 55.7 x2 + 102.6 x3
La función discriminante en la
frontera es
1713.2
– 34.9 x1 - 28 x2 - 51.3 x3 = 0
Ejemplo
6
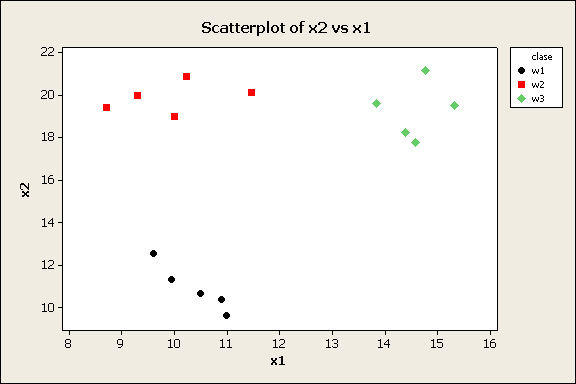
Se tiene información
correspondiente a tres clases en un espacio tridimensional, dada en la siguiente
tabla. Determinar para dicha información
|
|
w1 |
w2 |
w3 |
|||
|
n |
x1 |
x2 |
x1 |
x2 |
x1 |
x2 |
|
1 |
10.49 |
10.67 |
10.00 |
18.96 |
14.76 |
21.16 |
|
2 |
10.89 |
10.38 |
8.71 |
19.41 |
14.38 |
18.22 |
|
3 |
9.95 |
11.30 |
10.23 |
20.88 |
15.32 |
19.51 |
|
4 |
9.60 |
12.56 |
11.46 |
20.13 |
14.57 |
17.77 |
|
5 |
10.99 |
9.62 |
9.29 |
19.98 |
13.83 |
19.61 |
a) Graficar los datos
correspondientes a las tres clases
b) Calcular las funciones discriminantes
para las tres clases
Results for: segmenta.MTW
Discriminant Analysis: clase versus x1, x2
Linear Method for Response: clase
Predictors: x1, x2
Group w1 w2 w3
Count 5 5 5
Summary of classification
True Group
Put into Group w1 w2
w3
w1 5 0
0
w2 0 5
0
w3 0 0
5
Total N 5 5
5
N correct 5
5 5
Proportion 1.000 1.000
1.000
N = 15 N Correct =
15 Proportion Correct = 1.000
Squared Distance Between Groups
w1 w2
w3
w1 0.0000 68.4786
98.5209
w2 68.4786 0.0000
37.1494
w3 98.5209 37.1494
0.0000
Linear Discriminant Function for Groups
w1 w2
w3
Constant -158.22 -278.50
-376.82
x1 19.38 19.63
27.65
x2 10.56 18.21
18.21
c) a que clase pertenece una observación dada por x
=[16.1, 17,4]T
f1 = -158.22 + 19.38 (16.1) + 10.56 (17.4) = 337.542
f2 = -278.50 + 19.63 (16.1) + 18.21 (17.4) = 354.397
f3 = -376.82
+ 27.65 (16.1) + 18.21 (17.4) = 385.199